千葉大学大学院医学研究院の星居孝之准教授、金田篤志教授らの研究チームは、急性骨髄性白血病細胞の中で働く遺伝子発現の新しい調節因子を発見しました。この新たに見いだしたDNA修復制御因子BOD1Lは、ヒストンメチル化酵素であるSETD1Aを直接的に制御する因子として作用していることが明らかとなりました。また、この2つの遺伝子の相互作用の破壊により、白血病細胞が死滅することも確認しました。さらに詳しい解析により、BOD1LはSETD1Aの古典的な役割であるヒストンメチル化機能とは異なる遺伝子発現調節機構を司ることによって、白血病細胞の生存に必須となることが明らかとなりました。このことからBOD1Lは、がんの新たな治療標的になると考えられます。
本研究成果は、科学誌Nucleic Acids Researchにて2024年7月12日(日本時間)にオンライン公開されました。
【本研究成果のポイント】
①急性骨髄性白血病は血液がんの一種で、骨髄中の白血球に分化する途中の未熟な細胞に異常が生じて、がん化した白血病細胞が異常に増える病気です。他の白血病に比べて進行が早く、再発の可能性も高いため、革新的な分子標的薬の開発が期待されています。
②SETD1Aというヒストンメチル化酵素は、がんの発生機構もしくは増殖機構において重要な役割を持つことが先行研究で明らかになってきていましたが、薬の標的となる場所が乏しく、治療への活用が困難でした。
③本研究の中でCRISPRタイリング法注1)やAIシステムなどの新しい技術の活用から見いだされたBOD1Lタンパク質の構造が、今後の創薬に結びつくことが期待されます。
■研究の背景
DNAの巻きつく核内タンパク質であるヒストンのメチル化は、遺伝子の発現制御に働くとされています。SETD1Aタンパク質は、原始的な真核生物である酵母のメチル化酵素と類似した構造を持つことから、ヒストンメチル化に必須であるとされてきました。一方で、研究チームは2018年に酵素活性とは独立した役割を持つことを発見しており、特有の役割が白血病細胞の生存に必要となることを報告していました参考資料1)。また2022年には、SETD1Aタンパク質を薬で分解するモデルを作り、白血病細胞の死滅や、ミトコンドリアの異常が生じることを報告していました参考資料2)。しかしながら、SETD1Aの働きを特異的に阻害する薬の開発はこれまで達成されておらず、治療への活用が困難でした。
■研究の成果
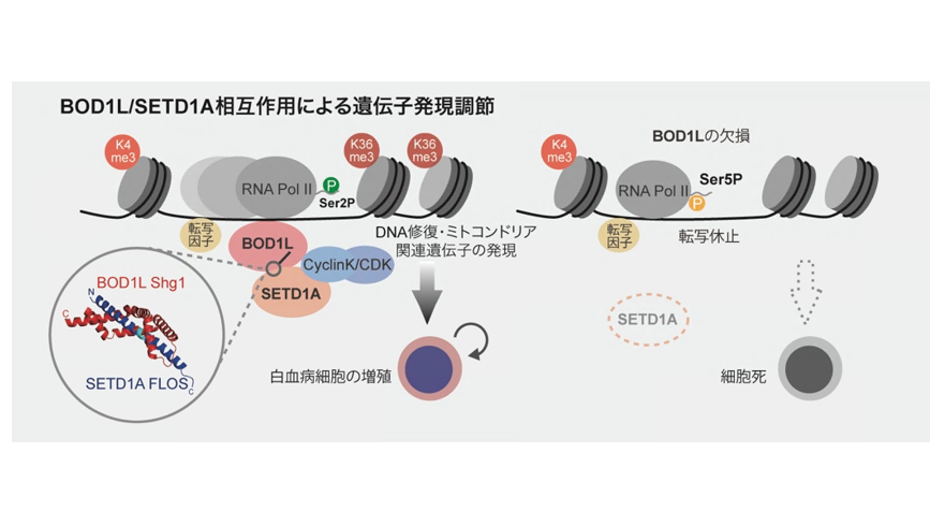
研究チームは、がんの細胞株を使って遺伝子の必要性を大規模に解析したDepMapデータベース注2)を基にして、SETD1Aと共依存性を示す分子を探索し、DNA修復制御因子BOD1Lを同定しました。BOD1Lは巨大なタンパク質で、詳細な機能解析は達成されていませんでしたが、本研究ではCRISPRタイリング法を活用して機能解析を実施し、白血病細胞の生存に必須となることを発見しました (図1)。
また、BOD1LとSETD1Aをそれぞれ欠損する白血病細胞で発現低下する遺伝子群はほぼ同一であり、BOD1L欠損時に観察される核内変化もSETD1Aを欠損した際の結果と同じでした。BOD1L欠損時には核内のDNA上からSETD1Aタンパク質が速やかに消失することが分かり、BOD1LはSETD1AのDNA結合の主導的な制御因子であることが分かりました。
BOD1LとSETD1Aの結合を詳しく調べると、2018年に発見したSETD1Aタンパク質上の機能性領域と、今回同定したBOD1L上の機能性領域が結合することが世界で初めて明らかになりました。AIシステムを使ったタンパク質複合体の予測から、2つのタンパク質が結びつく様子が予測され、研究チームの検証結果も構造予測の正確性を裏づけるものとなりました。また、BOD1Lタンパク質はSETD1Aタンパク質の一部に巻きつく特徴的な形をしており、新たな創薬標的として有用であることが示唆されました。
2つのタンパク質が結合した場合に限り、その周囲に特徴的な分子が存在することを予測し、split-TurboID注3)を活用して解析を行った結果から、多数の転写制御因子がこの複合体と連携して作用していることも分かりました。以上の結果から、BOD1LはSETD1Aと結合することにより、DNA修復制御因子としてよりも、がんの増殖や生存を助ける転写促進因子として働くことが明らかとなりました。
■研究者のコメント(千葉大学大学院医学研究院 星居准教授)
様々な技術の活用と研究者間の協力により、長らく不明であったタンパク質間相互作用の働きや構造の謎を解くことができたことは嬉しく思います。SETD1A自体は小児疾患や統合失調症の原因としても注目されているため、今回の発見ががん以外の疾患の治療にも広く貢献することを期待しています。
■用語解説
注1)CRISPRタイリング法:遺伝子編集技術CRISPR/Cas9で用いるsgRNAを一つの遺伝子に対して無数に設計し、タンパク質上の機能を持つ場所を網羅的に検査・同定する手法。
注2)DepMapデータベース:1,000以上のがん細胞株で行われた大規模CRISPR-Cas9スクリーニング結果などから、がんの治療標的の発見や治療法開発を促すことを目的としたデータベース。
注3)split-TurboID:近接依存性標識法の一つで、2つのタンパク質が相互作用した時に限定して、その周囲に存在するタンパク質を標識し、同定することができる。
■論文情報
論文タイトル:BOD1L mediates chromatin binding and non-canonical function of H3K4 methyltransferase SETD1A
著者:Takayuki Hoshii*, Sota Kikuchi, Tomoya Kujirai, Takeshi Masuda, Tomoko Ito, Satoshi Yasuda, Makoto Matsumoto, Bahityar Rahmutulla, Masaki Fukuyo, Takeshi Murata, Hitoshi Kurumizaka, Atsushi Kaneda *責任著者
雑誌名:Nucleic Acids Research
DOI:10.1093/nar/gkae605
■参考資料1
論文タイトル:A Non-catalytic function of SETD1A regulates Cyclin K and the DNA damage response
雑誌名:Cell
DOI:10.1016/j.cell.2018.01.032
■参考資料2
論文タイトル:SETD1A regulates transcriptional pause release of heme biosynthesis genes in leukemia
雑誌名:Cell Reports
DOI:10.1016/j.celrep.2022.111727
■研究プロジェクトについて
本研究は、科研費(19H03690, 22H03099, 22H04684, 22K19538, 20H05690, 22K15033, 23K17392, 23H05475)、武田科学振興財団、ノバルティス科学振興財団、小林がん学術振興会、上原記念生命科学財団、AMED P-PROMOTE(23ama221118)、AMED BINDS(23ama121018, 23ama121013, 23ama121009)、ERATO(JPMJER1901)、千葉大学国際高等研究基幹の支援を受けて行われました。









